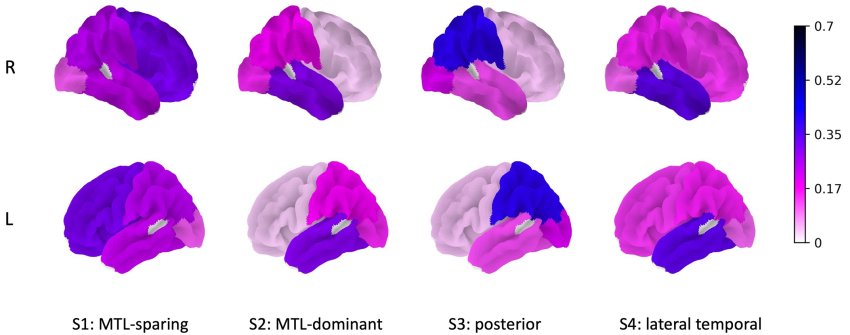
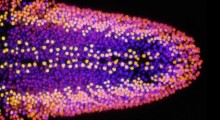
 Grafici delle superfici cerebrali degli emisferi destro e sinistro, che mostrano la forza del vettore canonico (νƙ) per ogni sottotipo. Fonte: Yang et al.
Grafici delle superfici cerebrali degli emisferi destro e sinistro, che mostrano la forza del vettore canonico (νƙ) per ogni sottotipo. Fonte: Yang et al.
Una nuova tecnica computazionale, che combina i dati genomici e di scansioni PET tau, promette un approccio più personalizzato per sottotipizzare il morbo di Alzheimer (MA). Sulla base di un nuovo quadro di aggregazione che usa l'analisi di correlazione canonica sparsa (SCCA, sparse canonical correlation analysis), l'approccio integrato ha avuto successo nell'identificare quattro sottotipi del MA e i primi geni associati a ciascuno. La ricerca è stata presentata alla riunione annuale della Society of Nuclear Medicine and Molecular Imaging.
Il MA è un disturbo geneticamente complesso. Numerosi sottotipi della malattia sono stati identificati in base alle scansioni PET delle placche amiloidi e dei grovigli neurofibrillari tau, i segni distintivi patologici della malattia. Inoltre, studi recenti di associazione del genoma hanno identificato molti geni con un ruolo nello sviluppo della malattia.
"Identificando diversi sottotipi del MA con informazioni sia genomiche che da scansioni, i ricercatori potrebbero ottenere potenziali nuove intuizioni sulla biologia sottostante della malattia e sulla sua progressione", ha dichiarato Joyita Dutta PhD, prof.ssa associata nel Dipartimento di Ingegneria Biomedica all'Università del Massachusetts di Amherst. "Comprendere le associazioni genetiche specifiche per ciascun sottotipo potrebbe anche portare allo sviluppo di approcci terapeutici personalizzati in futuro".
I ricercatori hanno usato dati di scansione e genomici tratti dall'Alzheimer's Disease Neuroimaging Initiative, includendo tutti i partecipanti che avevano avuto una genotipizzazione con PET 18F-Flortaucipipir e Illumina SNP: 541 in totale, 334 cognitivamente normali e 207 deteriorati. Hanno calcolato i rapporti standardizzato del valore di assorbimento della PET tau di 10 regioni generali e hanno identificato ed estratto 145 variazioni del genoma associate al MA dalla genotipizzazione SNP. Hanno quindi applicato Il quadro di aggregazione SCCA congiuntamente ai set di dati PET tau e genomici.
Nell'analisi sono stati identificati quattro sottotipi di MA, come nell'immagine sopra:
- MTL-sparing (che risparmia l'MTL, il lobo temporale mediale),
- MTL-dominant (dominante nel lobo temporale mediale, o MTL),
- posterior (posteriore),
- lateral temporal (temporale laterale).
Oltre al gene ApoE, sono stati identificati anche i primi geni associati a ciascun sottotipo.
"La sottotipizzazione individualizzata via genomica e scansione è vitale per il MA, perché diversi sottotipi possono anche avere tassi e profili distinti di declino cognitivo, influenzando potenzialmente gli esiti di sperimentazione clinica e la risposta al trattamento", ha affermato la Dutta. “Combinando le informazioni di scansione molecolare con la genomica, abbiamo creato una tecnica diagnostica che potrebbe essere veramente personalizzata per ogni paziente. Ciò ha un potenziale per un'ampia utilità diagnostica in molti tipi di malattie, non solo il MA".
Fonte: Society of Nuclear Medicine and Molecular Imaging (> English) - Traduzione di Franco Pellizzari.
Riferimenti: F Yang, [+2], J Dutta. An SCCA-clustering framework for Alzheimer’s disease subtyping using tau PET and genomics. SNMI, 27 June 2023, Abstract 1377
Copyright: Tutti i diritti di testi o marchi inclusi nell'articolo sono riservati ai rispettivi proprietari.
Liberatoria: Questo articolo non propone terapie o diete; per qualsiasi modifica della propria cura o regime alimentare si consiglia di rivolgersi a un medico o dietologo. Il contenuto non rappresenta necessariamente l'opinione dell'Associazione Alzheimer OdV di Riese Pio X ma solo quella dell'autore citato come "Fonte". I siti terzi raggiungibili da eventuali collegamenti contenuti nell'articolo e/o dagli annunci pubblicitari sono completamente estranei all'Associazione, il loro accesso e uso è a discrezione dell'utente. Liberatoria completa qui.
Nota: L'articolo potrebbe riferire risultati di ricerche mediche, psicologiche, scientifiche o sportive che riflettono lo stato delle conoscenze raggiunte fino alla data della loro pubblicazione.







 Associazione Alzheimer OdV
Associazione Alzheimer OdV