 Ricercatori della Facoltà di Medicina della Università di Toronto (UofT) hanno scoperto una base genetica delle differenze fondamentali tra gli esseri umani e gli altri vertebrati, che potrebbe anche aiutare a spiegare perché gli esseri umani sono sensibili a malattie che non si trovano in altre specie.
Ricercatori della Facoltà di Medicina della Università di Toronto (UofT) hanno scoperto una base genetica delle differenze fondamentali tra gli esseri umani e gli altri vertebrati, che potrebbe anche aiutare a spiegare perché gli esseri umani sono sensibili a malattie che non si trovano in altre specie.
Gli scienziati si sono chiesti perché le specie di vertebrati, che sembrano e si comportano in modo molto diverso l'una dall'altra, però condividono repertori molto simili di geni, come, ad esempio, quelli presenti sia negli esseri umani che negli scimpanzè, nonostante le loro evidenti differenze fisiche.
Il team ha sequenziato e confrontato la composizione di centinaia di migliaia di messaggi genetici di organi equivalenti, come il cervello, il cuore e il fegato, provenienti da 10 diverse specie di vertebrati, che vanno dall'uomo alla rana. Essi hanno scoperto che lo splicing [= assemblaggio / saldatura / giunzione] alternativo - processo che permette a un singolo gene di dare origine a proteine diverse - ha cambiato radicalmente la struttura e la complessità dei messaggi genetici durante l'evoluzione dei vertebrati.
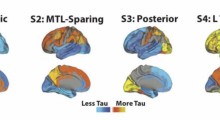
I risultati suggeriscono che le differenze nello splicing dei messaggi genetici hanno avuto un ruolo fondamentale nell'evoluzione delle caratteristiche fondamentali della specie. Tuttavia, lo stesso processo che fa sembrare diverse tra loro le specie potrebbe anche spiegare le differenze nella loro suscettibilità alle malattie. "Gli stessi meccanismi genetici responsabili dell'identità di una specie potrebbero aiutare gli scienziati a capire perché gli esseri umani sono soggetti a determinate malattie come l'Alzheimer e a particolari tipi di cancro che non si trovano in altre specie", dice Nuno Barbosa-Morais, autore principale dello studio, biologo computazionale del Donnelly Centre for Cellular and Biomolecular Research nella Facoltà di Medicina della UofT. "La nostra ricerca può portare a progettare approcci più efficaci per studiare e curare le malattie umane".
Una delle scoperte principali del team è che il processo di splicing alternativo è più complesso negli esseri umani e negli altri primati rispetto ad altre specie, come topi, polli e rane. "Le nostre osservazioni forniscono una nuova visione sulla base genetica della complessità di organi come il cervello umano", dice Benjamin Blencowe, Professore del Banting and Best Department of Research e del Dipartimento di Genetica Molecolare della UofT, nonchè autore senior dello studio.
"Il fatto che lo splicing alternativo sia molto differente anche tra le specie di vertebrati strettamente connesse, potrebbe in ultima analisi contribuire a spiegare perchè siamo unici".
***********************
Cosa pensi di questo articolo? Ti è stato utile? Hai rilievi, riserve, integrazioni? Conosci casi o ti è successo qualcosa che lo conferma? o lo smentisce? Puoi usare il modulo dei commenti qui sotto per dire la tua opinione. Che è importante e unica.
***********************
Fonte: Materiale della University of Toronto, via EurekAlert!, a service of AAAS.
Pubblicato in ScienceDaily il 20 Dicembre 2012 - Traduzione di Franco Pellizzari.
Copyright: Tutti i diritti di eventuali testi o marchi citati nell'articolo sono riservati ai rispettivi proprietari.
Liberatoria: Questo articolo non propone terapie o diete; per qualsiasi modifica della propria cura o regime alimentare si consiglia di rivolgersi a un medico o dietologo. Il contenuto non dipende da, nè impegna l'Associazione Alzheimer onlus di Riese Pio X. I siti terzi raggiungibili da eventuali links contenuti nell'articolo e/o dagli annunci pubblicitari proposti da Google sono completamente estranei all'Associazione, il loro accesso e uso è a discrezione dell'utente. Liberatoria completa qui.
Nota: L'articolo potrebbe riferire risultati di ricerche mediche, psicologiche, scientifiche o sportive che riflettono lo stato delle conoscenze raggiunte fino alla data della loro pubblicazione.
| Sostieni l'Associazione; una donazione, anche minima, ci aiuterà ad assistere malati e famiglie e continuare ad informarti. Clicca qui a destra: |







 Associazione Alzheimer OdV
Associazione Alzheimer OdV