|
Uno studio collaborativo pubblicato ieri sulla rivista Cell Reports fornisce l'evidenza di una nuova causa molecolare della neurodegenerazione provocata dal morbo di Alzheimer (MA).
Lo studio, condotto da ricercatori del Baylor College of Medicine e del Jan and Dan Duncan Neurological Research Institute al CrypSplice, integra i dati ottenuti da campioni autoptici del cervello umano e da moscerini della frutta per rivelare un legame meccanicistico innovativo tra le alterazioni dello splicing dell'RNA e la neurodegenerazione mediata dalla tau nel MA.
“Le cellule svolgono le loro funzioni producendo specifiche proteine codificate nei loro geni. Per produrre proteine, i geni codificati nel DNA vengono prima trascritti in molecole di RNA, che poi vengono tradotte in proteine”, ha detto l'autore senior dott. Joshua Shulman, professore associato di neurologia, neuroscienze e genetica molecolare ed umana al Baylor e ricercatore del Jan e Dan Duncan Neurological Research Institute.
In questo studio, Shulman e colleghi hanno studiato un meccanismo molecolare chiamato 'splicing dell'RNA' che è coinvolto nella produzione di molecole mature di RNA, necessarie per produrre proteine funzionanti. Hanno esaminato la possibilità che aggregati della proteina tau all'interno dei neuroni (un indicatore chiave del MA), interferiscano con lo splicing dell'RNA.
“Le alterazioni dello splicing dell'RNA sono note per essere coinvolte nello sviluppo di determinate condizioni neurodegenerative, come l'atrofia muscolare spinale e la sclerosi laterale amiotrofica. Tuttavia, fino ad ora, il loro ruolo nel MA non è stato studiato nei minimi dettagli“, ha detto Shulman.
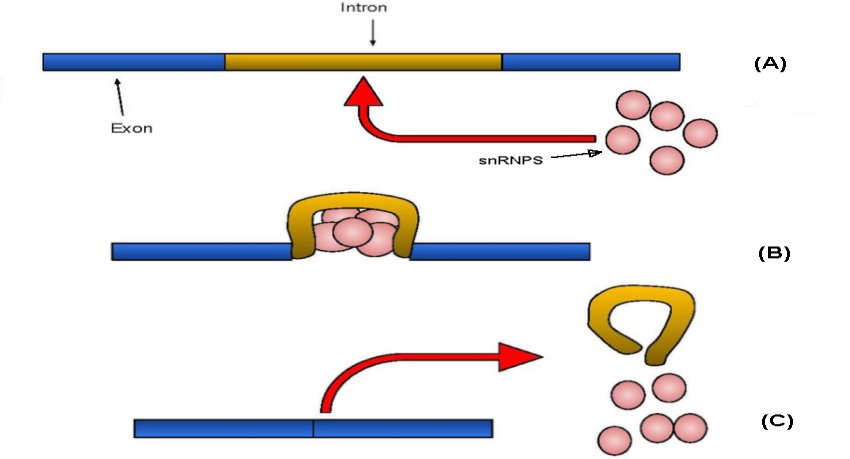
Per effettuare lo splicing dell'RNA, le cellule usano il complesso spliceosomale, un macchinario cellulare multiproteico che coordina la produzione di molecole mature di RNA. Lo splicing dell'RNA è uno dei modi con cui importanti organi generano differenti tipi di cellule, ciascuno dei quali ha funzioni specializzate ed è particolarmente critico per generare la diversità cellulare e la complessità nel cervello umano.
Si ritiene che perturbazioni minori nel montaggio e/o nel funzionamento dello spliceosoma rendano le cellule cerebrali vulnerabili alla degenerazione e alla morte prematura, soprattutto nei soggetti anziani.
Studi recenti su cervelli umani postmortem mostrano che diversi componenti del complesso spliceosomale possono legarsi e co-aggregarsi con i grovigli neurofibrillari tau. Nello studio attuale, i ricercatori hanno iniziato sovra-esprimendo tau tossica nei moscerini della frutta, come modello per verificare se le interazioni spliceosoma-tau possono causare una neurodegenerazione.
“Usando interazioni genetiche e test funzionali, Yi-Chen Hsieh e Caiwei Guo, ricercatori del mio laboratorio, hanno identificato diversi geni spliceosomali che mediano la tossicità tau”, ha detto Shulman.
Poiché molte proteine spliceosomali erano presenti a bassi livelli nei neuroni dei moscerini con sovra-espressione di tau tossica, i ricercatori hanno proposto che gli aggregati di tau interrompono l'assemblaggio corretto dello spliceosoma, oppure segregano dei componenti chiave nel citoplasma, lontano dal sito di azione nel nucleo.
Questo modello ha avuto ulteriore supporto da esperimenti in cui i moscerini esprimenti tau tossica hanno rivelato interruzioni globali nello splicing dell'RNA con il risultato di avere migliaia di RNA errate.
Analizzare gli errori di splicing specifici che si verificano a bassa frequenza in grandi serie di dati è un compito impegnativo che richiede molto tempo. Per condurre questa analisi, e automatizzare il processo, Shulman e i suoi colleghi hanno collaborato con scienziati computazionali del laboratorio del dott. Zhandong Liu, professore associato di pediatria-neurologia del Baylor e anch'egli ricercatore del Jan and Dan Duncan Neurological Research Institute.
“Identificare e classificare gli errori di splicing specifici si è rivelato un compito abbastanza difficile. Gli strumenti di calcolo attuali, che analizzano set di dati di sequenziamento dell'RNA, di solito filtrano qualsiasi modifica che non corrisponde ai normali schemi di splicing dell'RNA. Abbiamo dovuto esaminare in modo specifico questo ‘materiale spazzatura’ per identificare i modelli di errori specifici di splicing“, ha detto Liu.
Per questo studio, il dottor Hari Krishna Yalamanchili, post-dottorato del laboratorio di Liu, ha modificato il CrypSplice, uno strumento innovativo di calcolo che aveva sviluppato in precedenza, per effettuare l'analisi approfondita degli errori di splicing, scoprendo un legame tra grovigli tau e un aumento in un particolare tipo di errori di splicing.
“Questo è il primo studio a dimostrare il ruolo degli aggregati tau nella rottura della localizzazione cellulare e della funzione dei componenti complessi spliceosomali, che si traduce in errori globali di splicing e causao la perdita progressiva di neuroni”, ha detto Shulman. “I nostri risultati aprono una possibilità nuova ed entusiasmante di usare lo splicing dell'RNA come bersaglio molecolare potenziale per il MA e per altre malattie neurodegenerative mediate dalla tau”.
|









 Associazione Alzheimer OdV
Associazione Alzheimer OdV